PEPATAC是基于python开发的一个ATAC数据分析的pipeline, 网址如下
http://code.databio.org/PEPATAC/
提供了从原始fastq到peak calling的分析功能,最终结果以html的形式呈现,方便查看,一个示例报告链接如下
http://code.databio.org/PEPATAC/files/examples/gold/summary.html
可以从以下3个方面来查看分析的结果
- Samples, 以样本为单位进行查看,给出了fastqc质量评估的结果,序列数,mapping比例,线粒体比例,peak个数等统计指标,以及插入片段长度分布,TSS富集分布等常用图表
- Objects, 这里表示分析内容的意思,用于查看感兴趣的分析内容,和单个样本不同的是,这里将所有样本放在一起,方便比较
- Summary, 汇总分析,将所有分析内容,所有样本的结果放在一起进行查看
该流程的可取之处在于其丰富的统计指标和可视化图表,部分图表展示如下
1. TSS enrichment plot
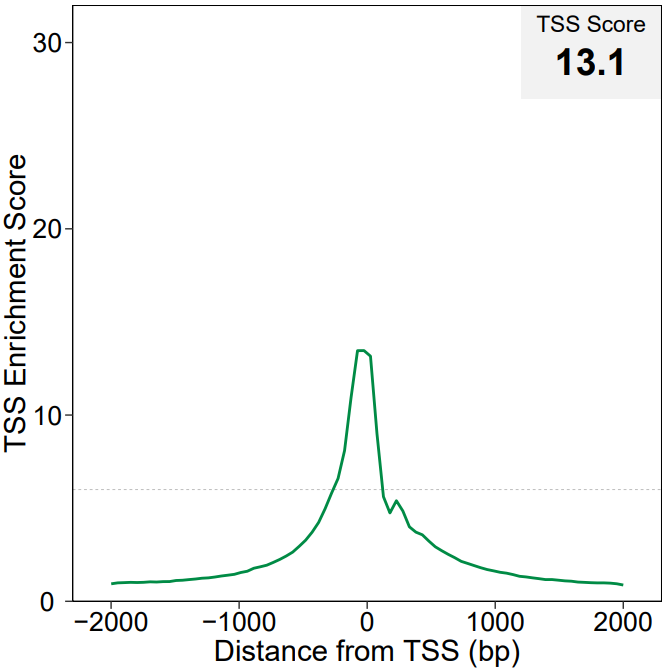
不仅给出了TSS上下游2kb的reads分布,还给出了TSS Enrichment Score。
2. TSS distance distribution
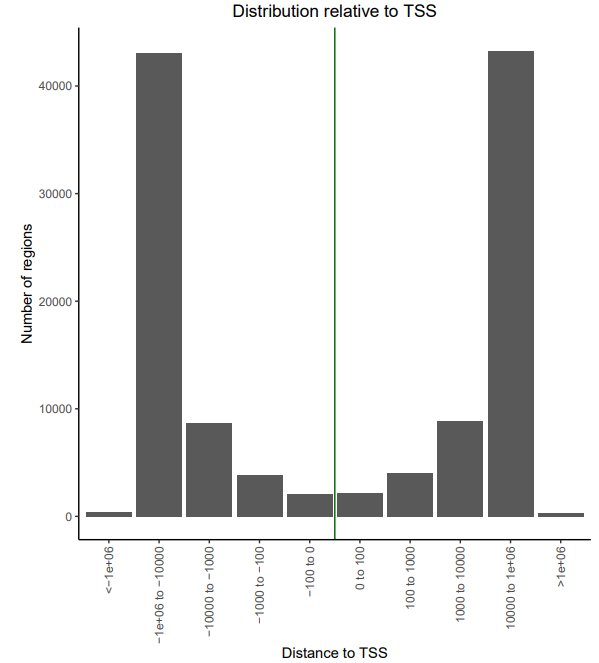
peak与TSS位点距离分布的直方图, 从图中可以看出,绝大多数的peak与TSS位点的距离在10kb到1000kb的一个区间。
3. Fragment length distribution
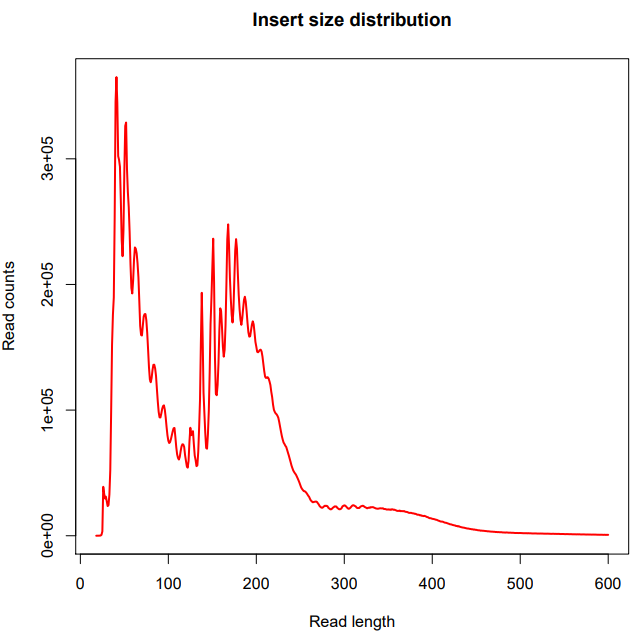
插入片段长度分布图,小于100bp的峰为NFR reads, 100到200bp之前的峰对应mononucleosome。
4. Peak chromsomal distribution

peak在染色体上的分布图,实际上就是一个区间分布的直方图,只不过这里的区间是染色体。
5. Peak genomic parition distribution

peak在各种基因组元件中分布的柱状图,如果peak与某个元件存在overlap, 就将对应的计数加1。可以看到,大部分的peak落在基因间区和内含子区。
6. Fraction of reads and/or peaks in features(FRiF)
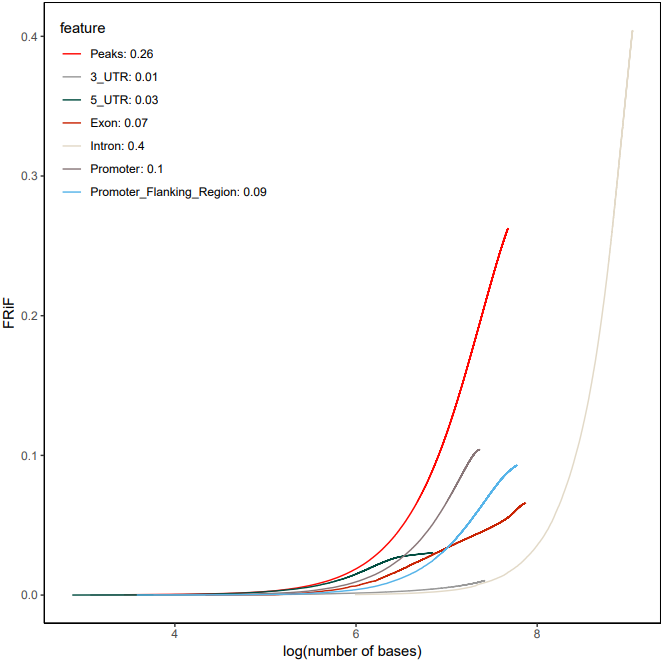
FRiP score的累积分布图,通过对bases进行抽样,计算不同抽样条件下的FRip score值,并绘制上图,类似饱和度分析。在这里,对FRip的概念进一步扩展,从原本定义中的peak区域扩展到了各种基因组元件中。
其丰富的统计指标也是该流程的一个亮点,有40个左右的统计指标,由于数量太多,这里就不展示了。查看上述demo的链接,可以看到更加详细的信息,关于软件的具体用请查看官方的帮助文档。
·end·

一个只分享干货的
生信公众号










