欢迎关注"生信修炼手册"!
breakdancer 是一款结构变异检测软件, 专门针对双端测序数据进行开发,github地址如下
https://github.com/genome/breakdancer
分析原理图如下
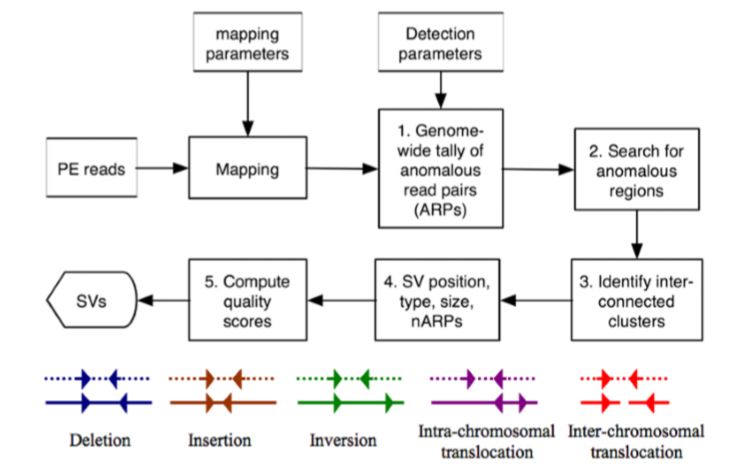
从原理图可以看出,breakdancer 会根据双端reads的比对情况,检测以下5种类型的结构变异
- insertions
- deletions
- inversions
- inter-chromosomal translocations
- intra-chromosomal translocations
软件的安装过程如下
git clone --recursive https://github.com/genome/breakdancer.git
cd breakdancer
mkdir build
cd build
cmake .. -DCMAKE_BUILD_TYPE=release -DCMAKE_INSTALL_PREFIX=/usr/local
make最终会生成一个可执行的二进制文件,breakdancer-max。软件的使用也比较简单,共两个步骤。
1. 生成配置文件
输入文件为比对基因组产生的bam文件, 用法如下
bam2cfg.pl tumor.bam normal.bam > config.txt配置文件中,每个样本对应一行记录,包含以下特征值
readgroup:tumor
platform:illumina
map:tumor.bam
readlen:144.84
lib:YL
num:10000
lower:0.00
upper:519.05
mean:210.24
std:65.87
SWnormality:-40.54
exe:samtools view2. 鉴定结构变异
用法如下
breakdancer_max -t -q 10 -d sv.reads config.txt > sv.out结构变异的检测计算量较大,所以需要的时间也很久。输出文件的列数很多,共有14列。
每一列的含义如下
- Chromosome 1
- Position 1
- Orientation 1
- Chromosome 2
- Position 2
- Orientation 2
- Type of a SV
- Size of a SV
- Confidence Score
- Total number of supporting read pairs
- Total number of supporting read pairs from each map file
- Estimated allele frequency
- Software version
- The run parameters
1到6列描述的是断裂点的位置信息;第7列描述结构变异的类型,DEL代表缺失,INS代表插入,INV代表倒位,ITX代表同一染色体上的易位,CTX代表不同染色体之间的易位;第8列代表结构变异的长度,对于染色体间的易位,这个数值没有含义;第9列代表该结构变异可信度的打分值,数值越大,可靠性越高。
扫描关注微信号,更多精彩内容等着你!










