1.技术背景&赛题介绍:
A Labeled Chinese Dataset for Diabetes中文糖尿病标注数据集详情请见。
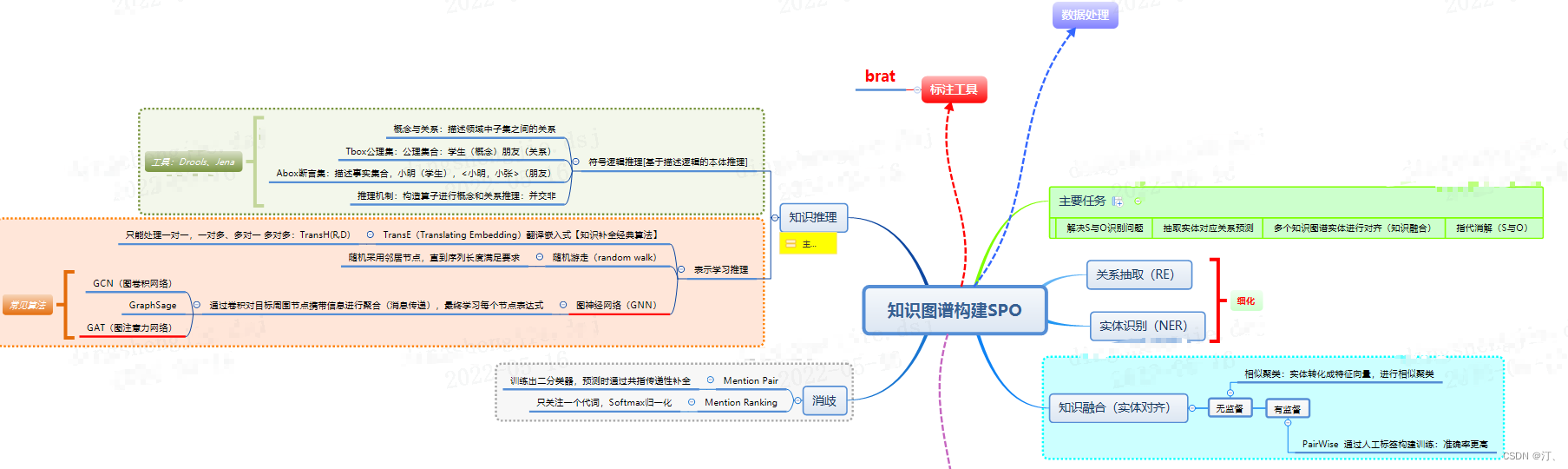
数据集链接:瑞金医院MMC人工智能辅助构建知识数据源:知识图谱构建SPO,知识图谱构建SPO-机器学习文档类资源-CSDN下载
代码链接:瑞金医院MMC人工智能辅助构建知识代码-机器学习文档类资源-CSDN下载
本地代码推荐BiLSTM+CRF(经调试效果佳):知识图谱项目实战(一):瑞金医院MMC人工智能辅助构建知识图谱--初赛实体识别【1】码源。-深度学习文档类资源-CSDN下载
赛题说明
本次大赛旨在通过糖尿病相关的教科书、研究论文来做糖尿病文献挖掘并构建糖尿病知识图谱。参赛选手需要设计高准确率,高效的算法来挑战这一科学难题。第一赛季课题为“基于糖尿病临床指南和研究论文的实体标注构建”,第二赛季课题为“基于糖尿病临床指南和研究论文的实体间关系构建”。本次大赛禁止使用外部数据,可以使用外部工具。本次大赛禁止通过构造字典方式来进行实体预测。
文件标注工作基于brat软件,brat rapid annotation tool。其中.txt文件为原始文档,.ann文件为标注信息,标注实体以T开头,后接实体序号,实体类别,起始位置和实体对应的文档中的词。如果需要在brat软件中查看标注结果,需要添加.conf文件。
初赛
提供与糖尿病相关的学术论文以及糖尿病临床指南,要求选手在学术论文和临床指南的基础上,做实体的标注。实体类别共十五类。
类别名称和定义
疾病相关:
1、疾病名称 (Disease),如I型糖尿病。
2、病因(Reason),疾病的成因、危险因素及机制。比如“糖尿病是由于胰岛素抵抗导致”,胰岛素抵抗是属于病因。
3、临床表现 (Symptom),包括症状、体征,病人直接表现出来的和需要医生进行查体得出来的判断。如"头晕" "便血" 等。
4、检查方法(Test),包括实验室检查方法,影像学检查方法,辅助试验,对于疾病有诊断及鉴别意义的项目等,如甘油三酯。
5、检查指标值(Test_Value),指标的具体数值,阴性阳性,有无,增减,高低等,如”>11.3 mmol/L”。
治疗相关:
6、药品名称(Drug),包括常规用药及化疗用药,比如胰岛素。
7、用药频率(Frequency),包括用药的频率和症状的频率,比如一天两次。
8、用药剂量(Amount),比如500mg/d。
9、用药方法(Method):比如早晚,餐前餐后,口服,静脉注射,吸入等。
10、非药治疗(Treatment),在医院环境下进行的非药物性治疗,包括放疗,中医治疗方法等,比如推拿、按摩、针灸、理疗,不包括饮食、运动、营养等。
11、手术(Operation),包括手术名称,如代谢手术等。
12、不良反应(SideEff),用药后的不良反应。
常规实体:
13、部位(Anatomy),包括解剖部位和生物组织,比如人体各个部位和器官,胰岛细胞。
14、程度(level),包括病情严重程度,治疗后缓解程度等。
15、持续时间(Duration),包括症状持续时间,用药持续时间,如“头晕一周”的“一周”。
复赛
提供与糖尿病相关的学术论文以及糖尿病临床指南。选手从中抽取实体之间的关系。实体之间关系共十类。
实体关系类别名称
1、检查方法 -> 疾病(Test_Disease)
2、临床表现 -> 疾病(Symptom_Disease)
3、非药治疗 -> 疾病(Treatment_Disease)
4、药品名称 -> 疾病(Drug_Disease)
5、部位 -> 疾病(Anatomy_Disease)
6、用药频率 -> 药品名称(Frequency_Drug)
7、持续时间 -> 药品名称(Duration_Drug)
8、用药剂量 -> 药品名称(Amount_Drug)
9、用药方法 -> 药品名称(Method_Drug)
10、不良反应 -> 药品名称(SideEff-Drug)
评估标准
采用F1-Measure作为评测指标。
复赛评测采用严格交集的方式来计算F1,即选手提交文件中的关系部分的第二列的整个字符串必须与答案完全一致。
选手提交格式
初赛提交结果为zip文件,参考submit。zip中的文件需要与测试txt文件的文件名相同,后缀名为.ann。文件中每一列以tab分割,共三列:第一列为实体编号,编号自拟且需唯一,不参与评测;第二列包含实体类别和实体的起始和终止位置,以空格分割,注意部分实体可能在第二列有分号,表示该实体跨行;第三列是实体所对应的词语, 不参与评测 。
复赛提交结果为zip文件,参考submit。zip中的文件名需要与测试的ann的文件名一致,并且保留测试ann文件的原有内容。在原有内容的基础上,后面添加行代表关系。关系行以tab分割,共两列:第一列为关系编号,以字符R开头,如“R1”,编号需唯一;第二列包含关系类别和关系的起始(以Arg1:开始,后接实体id,如“Arg1:T1”)和终止位置(以Arg2:开始,后接实体id,如“Arg2:T2”),以空格分割。
2.数据预处理
数据连接见文章开头
把数据解压最好放到同路径下即可
! unzip data.zip 去终端cd 路径 解压即可安装pandas库后
pip install pandasimport pandas as pd
label=pd.read_csv("train/0.ann",header=None,sep="\t")
print(label)0 1 2
0 T1 Disease 1845 1850 1型糖尿病
1 T2 Disease 1983 1988 1型糖尿病
2 T4 Disease 30 35 2型糖尿病
3 T5 Disease 1822 1827 2型糖尿病
4 T6 Disease 2055 2060 2型糖尿病
.. ... ... ...
593 R206 Symptom_Disease Arg1:T329 Arg2:T325 NaN
594 R207 Symptom_Disease Arg1:T331 Arg2:T325 NaN
595 R208 Test_Disease Arg1:T337 Arg2:T338 NaN
596 R209 Treatment_Disease Arg1:T343 Arg2:T345 NaN
597 R210 Treatment_Disease Arg1:T344 Arg2:T345 NaN
[598 rows x 3 columns]entity&connect的标注格式化解析
label_T=label[label[0].str.startswith("T")] #选出开头
0 1 2
0 T1 Disease 1845 1850 1型糖尿病
1 T2 Disease 1983 1988 1型糖尿病
2 T4 Disease 30 35 2型糖尿病
3 T5 Disease 1822 1827 2型糖尿病
4 T6 Disease 2055 2060 2型糖尿病
.. ... ... ...
383 T309 Disease 5984 5987 糖尿病
384 T310 Test 6335 6339 血红蛋白
385 T385 Test 6340 6345 红细胞转换
386 T343 Disease 6616 6621 2型糖尿病
387 T344 Test 6621 6629 HBA1C c
[388 rows x 3 columns]
label_T.columns=["id","entity","text"] #表头设置,并划分信息
label_T["category"]=[e.split()[0] for e in label_T["entity"].tolist()]
label_T["start"]=[int(e.split()[1]) for e in label_T["entity"].tolist()]
label_T["end"]=[int(e.split()[-1]) for e in label_T["entity"].tolist()]
print(label_T)
id entity text category start end
0 T1 Disease 1845 1850 1型糖尿病 Disease 1845 1850
1 T2 Disease 1983 1988 1型糖尿病 Disease 1983 1988
2 T4 Disease 30 35 2型糖尿病 Disease 30 35
3 T5 Disease 1822 1827 2型糖尿病 Disease 1822 1827
4 T6 Disease 2055 2060 2型糖尿病 Disease 2055 2060
.. ... ... ... ... ... ...
383 T309 Disease 5984 5987 糖尿病 Disease 5984 5987
384 T310 Test 6335 6339 血红蛋白 Test 6335 6339
385 T385 Test 6340 6345 红细胞转换 Test 6340 6345
386 T343 Disease 6616 6621 2型糖尿病 Disease 6616 6621
387 T344 Test 6621 6629 HBA1C c Test 6621 6629
[388 rows x 6 columns]同理对待实体之间的关系:
label_R=label[label[0].str.startswith("R")] #选出开头
print(label_R)
0 1 2
388 R1 Test_Disease Arg1:T369 Arg2:T368 NaN
389 R2 Test_Disease Arg1:T13 Arg2:T25 NaN
390 R3 Test_Disease Arg1:T14 Arg2:T25 NaN
391 R4 Test_Disease Arg1:T3 Arg2:T25 NaN
392 R5 Test_Disease Arg1:T31 Arg2:T33 NaN
.. ... ... ...
593 R206 Symptom_Disease Arg1:T329 Arg2:T325 NaN
594 R207 Symptom_Disease Arg1:T331 Arg2:T325 NaN
595 R208 Test_Disease Arg1:T337 Arg2:T338 NaN
596 R209 Treatment_Disease Arg1:T343 Arg2:T345 NaN
597 R210 Treatment_Disease Arg1:T344 Arg2:T345 NaN
[210 rows x 3 columns]
label_R.columns=["id","relation","nan"] #表头设置,并划分信息
label_R["category"]=[r.split()[0] for r in label_R["relation"].tolist()]
label_R["arg1"]=[r.split()[1][5:] for r in label_R["relation"].tolist()]
label_R["arg2"]=[r.split()[2][5:] for r in label_R["relation"].tolist()]
print(label_R)
id relation nan category \
388 R1 Test_Disease Arg1:T369 Arg2:T368 NaN Test_Disease
389 R2 Test_Disease Arg1:T13 Arg2:T25 NaN Test_Disease
390 R3 Test_Disease Arg1:T14 Arg2:T25 NaN Test_Disease
391 R4 Test_Disease Arg1:T3 Arg2:T25 NaN Test_Disease
392 R5 Test_Disease Arg1:T31 Arg2:T33 NaN Test_Disease
.. ... ... ... ...
593 R206 Symptom_Disease Arg1:T329 Arg2:T325 NaN Symptom_Disease
594 R207 Symptom_Disease Arg1:T331 Arg2:T325 NaN Symptom_Disease
595 R208 Test_Disease Arg1:T337 Arg2:T338 NaN Test_Disease
596 R209 Treatment_Disease Arg1:T343 Arg2:T345 NaN Treatment_Disease
597 R210 Treatment_Disease Arg1:T344 Arg2:T345 NaN Treatment_Disease
arg1 arg2
388 T369 T368
389 T13 T25
390 T14 T25
391 T3 T25
392 T31 T33
.. ... ...
593 T329 T325
594 T331 T325
595 T337 T338
596 T343 T345
597 T344 T345
[210 rows x 6 columns]后续会讲把里面处理过的冗余信息提出
3.初赛--实体识别
本地代码推荐BiLSTM+CRF(经调试效果佳):瑞金医院MMC人工智能辅助构建知识代码-机器学习文档类资源-CSDN下载
下面给出常见方法的核心代码,使用方法只需要把上面BiLSTM+CRF对应算法部分替换即可
3.1 传统方法:概率图模型——条件随机场CRF
# 构建概率图模型——条件随机场
import keras
from keras.layers import Input, Embedding
from keras_contrib.layers import CRF
from keras.models import Model
def build_crf_model(num_cates, seq_len, vocab_size, model_opts=dict()):
opts = {
'emb_size': 256,
'emb_trainable': True,
'emb_matrix': None,
'optimizer': keras.optimizers.Adam()
}
opts.update(model_opts)
input_seq = Input(shape=(seq_len,), dtype='int32')
if opts.get('emb_matrix') is not None:
embedding = Embedding(vocab_size, opts['emb_size'],
weights=[opts['emb_matrix']],
trainable=opts['emb_trainable'])
else:
embedding = Embedding(vocab_size, opts['emb_size'])
x = embedding(input_seq)
crf = CRF(num_cates, sparse_target=True)
output = crf(x)
model = Model(input_seq, output)
model.compile(opts['optimizer'], loss=crf.loss_function, metrics=[crf.accuracy])
return model# CRF条件随机场实例化
seq_len = sent_len + 2 * sent_pad
model = build_crf_model(num_cates, seq_len=seq_len, vocab_size=vocab_size,model_opts={'emb_matrix': w2v_embeddings, 'emb_size': 100, 'emb_trainable': False})
model.summary()
# 条件随机场模型训练
model.fit(train_X, train_y, batch_size=64, epochs=10)效果展示:
Epoch 8/10
2622/2622 [==============================] - 6s 2ms/step - loss: 0.7750 - crf_viterbi_accuracy: 0.7705
Epoch 9/10
2622/2622 [==============================] - 5s 2ms/step - loss: 0.7346 - crf_viterbi_accuracy: 0.7745
Epoch 10/10
2622/2622 [==============================] - 5s 2ms/step - loss: 0.7014 - crf_viterbi_accuracy: 0.7773# 输出评价指标
f_score, precision, recall = Evaluator.f1_score(test_docs, pred_docs)
print('f_score: ', f_score)
print('precision: ', precision)
print('recall: ', recall)
f_score: 0.4847994451508496
precision: 0.5801632314289666
recall: 0.416360567854660973.2 深度学习基础算法-RNN模型加crf模型
# 构建RNN模型加crf模型
import keras
from keras.layers import Input, SimpleRNN, Embedding, Bidirectional
from keras_contrib.layers import CRF
from keras.models import Model
def build_rnn_model(num_cates, seq_len, vocab_size, model_opts=dict()):
opts = {
'emb_size': 256,
'emb_trainable': True,
'emb_matrix': None,
'rnn_units': 256,
'optimizer': keras.optimizers.Adam()
}
opts.update(model_opts)
input_seq = Input(shape=(seq_len,), dtype='int32')
if opts.get('emb_matrix') is not None:
embedding = Embedding(vocab_size, opts['emb_size'],
weights=[opts['emb_matrix']],
trainable=opts['emb_trainable'])
else:
embedding = Embedding(vocab_size, opts['emb_size'])
x = embedding(input_seq)
rnn = SimpleRNN(opts['rnn_units'], return_sequences=True)
x = rnn(x)
crf = CRF(num_cates, sparse_target=True)
output = crf(x)
model = Model(input_seq, output)
model.compile(opts['optimizer'], loss=crf.loss_function, metrics=[crf.accuracy])
return modelEpoch 7/10
2622/2622 [==============================] - 15s 6ms/step - loss: 0.4254 - crf_viterbi_accuracy: 0.8507
Epoch 8/10
2622/2622 [==============================] - 15s 6ms/step - loss: 0.4047 - crf_viterbi_accuracy: 0.8560
Epoch 9/10
2622/2622 [==============================] - 15s 6ms/step - loss: 0.3860 - crf_viterbi_accuracy: 0.8605
Epoch 10/10
2622/2622 [==============================] - 15s 6ms/step - loss: 0.3729 - crf_viterbi_accuracy: 0.8625
f_score: 0.692128403432567
precision: 0.7419647927314026
recall: 0.6485654720540057性能已经提升了很多了。
3.3 深度学习高级算法--LSTM加crf模型
# 构建长短时记忆模型模型加crf模型
import keras
from keras.layers import Input, LSTM, Embedding, Bidirectional
from keras_contrib.layers import CRF
from keras.models import Model
def build_lstm_model(num_cates, seq_len, vocab_size, model_opts=dict()):
opts = {
'emb_size': 256,
'emb_trainable': True,
'emb_matrix': None,
'rnn_units': 256,
'optimizer': keras.optimizers.Adam()
}
opts.update(model_opts)
input_seq = Input(shape=(seq_len,), dtype='int32')
if opts.get('emb_matrix') is not None:
embedding = Embedding(vocab_size, opts['emb_size'],
weights=[opts['emb_matrix']],
trainable=opts['emb_trainable'])
else:
embedding = Embedding(vocab_size, opts['emb_size'])
x = embedding(input_seq)
lstm = LSTM(opts['rnn_units'], return_sequences=True)
x = lstm(x)
crf = CRF(num_cates, sparse_target=True)
output = crf(x)
model = Model(input_seq, output)
model.compile(opts['optimizer'], loss=crf.loss_function, metrics=[crf.accuracy])
return model2622/2622 [==============================] - 37s 14ms/step - loss: 0.4122 - crf_viterbi_accuracy: 0.8570
Epoch 7/10
2622/2622 [==============================] - 37s 14ms/step - loss: 0.3836 - crf_viterbi_accuracy: 0.8642
Epoch 8/10
2622/2622 [==============================] - 37s 14ms/step - loss: 0.3598 - crf_viterbi_accuracy: 0.8708
Epoch 9/10
2622/2622 [==============================] - 37s 14ms/step - loss: 0.3386 - crf_viterbi_accuracy: 0.8758
Epoch 10/10
2622/2622 [==============================] - 36s 14ms/step - loss: 0.3150 - crf_viterbi_accuracy: 0.8816f_score: 0.6853306967788938
precision: 0.7201443569553806
recall: 0.65372778715377743.4 主流算法:BiLSTM+CRF
# 构建双向长短时记忆模型模型加crf模型
import keras
from keras.layers import Input, LSTM, Embedding, Bidirectional
from keras_contrib.layers import CRF
from keras.models import Model
def build_lstm_crf_model(num_cates, seq_len, vocab_size, model_opts=dict()):
opts = {
'emb_size': 256,
'emb_trainable': True,
'emb_matrix': None,
'lstm_units': 256,
'optimizer': keras.optimizers.Adam()
}
opts.update(model_opts)
input_seq = Input(shape=(seq_len,), dtype='int32')
if opts.get('emb_matrix') is not None:
embedding = Embedding(vocab_size, opts['emb_size'],
weights=[opts['emb_matrix']],
trainable=opts['emb_trainable'])
else:
embedding = Embedding(vocab_size, opts['emb_size'])
x = embedding(input_seq)
lstm = LSTM(opts['lstm_units'], return_sequences=True)
x = Bidirectional(lstm)(x)
crf = CRF(num_cates, sparse_target=True)
output = crf(x)
model = Model(input_seq, output)
model.compile(opts['optimizer'], loss=crf.loss_function, metrics=[crf.accuracy])
return model完整代码见前面,下载即可。
Epoch 8/10
2622/2622 [==============================] - 88s 34ms/step - loss: 0.2898 - crf_viterbi_accuracy: 0.9190
Epoch 9/10
2622/2622 [==============================] - 88s 33ms/step - loss: 0.2664 - crf_viterbi_accuracy: 0.9248
Epoch 10/10
2622/2622 [==============================] - 87s 33ms/step - loss: 0.2430 - crf_viterbi_accuracy: 0.9313f_score: 0.7060570071258908
precision: 0.7038973852984707
recall: 0.7082299215725206性能显著提升。
看一下可视化效果:标注出来的实体还可以

4.小结
后续将会分享实体标注的其他算法和开源工具以及关系抽取的实现









