欢迎关注"生信修炼手册"!
对于GWAS分析的结果,最常见的可视化手段就是曼哈顿图了。一个典型的曼哈顿图示例如下
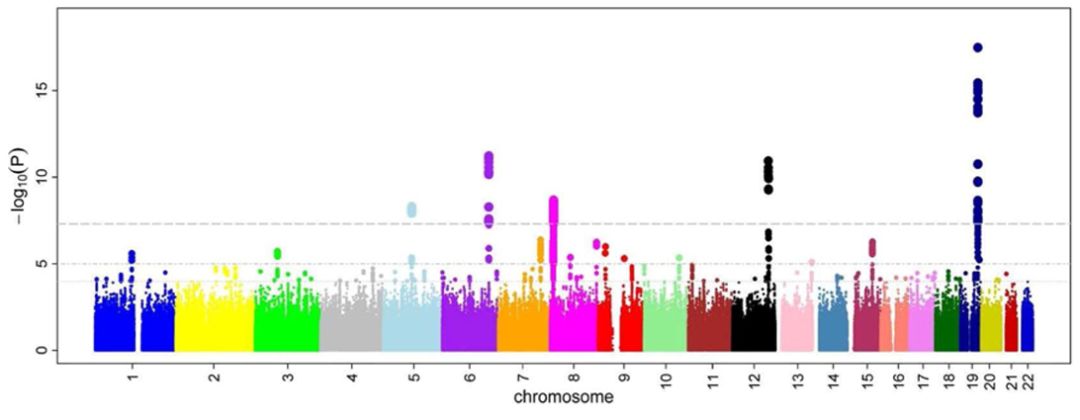
图中的每个点代表一个SNP位点,横坐标是SNP位点在染色体上的位置,纵坐标是关联分析计算出的p值。为了美观,不同染色体上的SNP位点用不同颜色标识。纵轴上的虚线代表阈值,p值越小,对应到纵轴的值越大。通常在阈值上方的SNP位点认为是和疾病或者形状相关联的位点。
曼哈顿图本质上就是一个散点图,绘制方法比较简单,有很多的R包可以直接出图,本篇主要介绍haploview软件。 这个软件可以直接读取plink关联分析的结果,然后绘制曼哈顿图。
在之前的文章中介绍了plink关联分析的方法,根据输入的hapmap1.ped和hapmap1.map两个文件,最终可以得到一个allel水平的关联分析结果,文件名为as1.assoc。haploview需要读取hapmap1.map和as1.assoc两个文件。
1. 导入数据
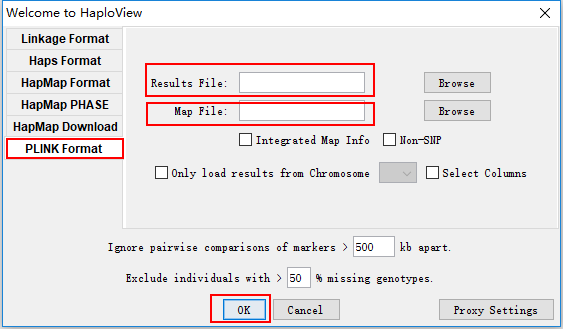
启动haploview之后,选择PLINK Format选项,Results File 选择关联分析的结果文件,可以是allel关联分析的后缀为.assoc的文件,也可以是包含了genotype关联分析的后缀为.model的文件;Map file 选择后缀为.map的文件,当然也可以选择后缀为.bim的二进制的map文件,选择好之后,点击OK即可。
2. 筛选数据
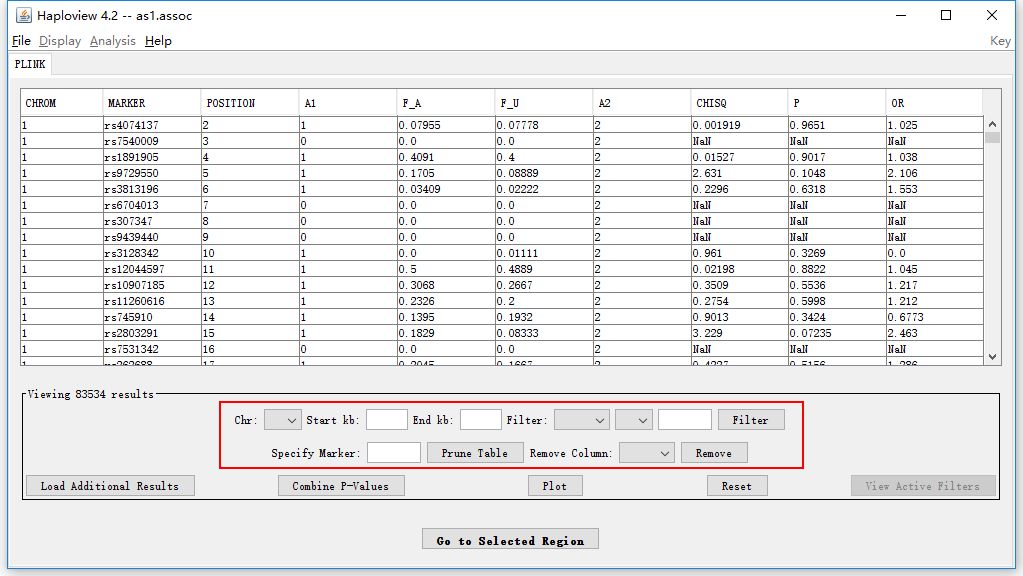
导入成功之后,可以看到如下所示的表格,这个表格里就是导入的关联分析的结果。红色方框标识的区域可以对关联分析的结果进行筛选,可以通过Chr, Start Kb, End kb对染色体区域进行筛选,也可以通过Filter对符合要求的SNP位点进行筛选。
3. 绘图
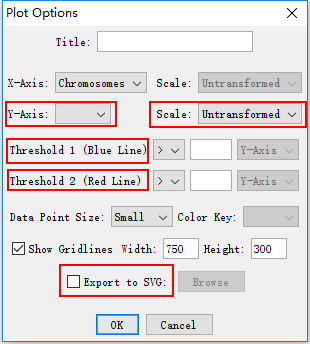
筛选好之后,点击Plot按钮,设置绘图的参数。X轴肯定是Chromosomes, Y轴通常选择P值,如果你有进行多重假设检验的校正,也可以选择校正之后的P值。scale表示对y轴的值进行处理,有一下3种处理
- untransformed
- -log10
- ln
一般情况下选择-log10。Export to SVG可以导出图片,格式为svg。
设置好之后,点击OK就可以出图了。示例如下
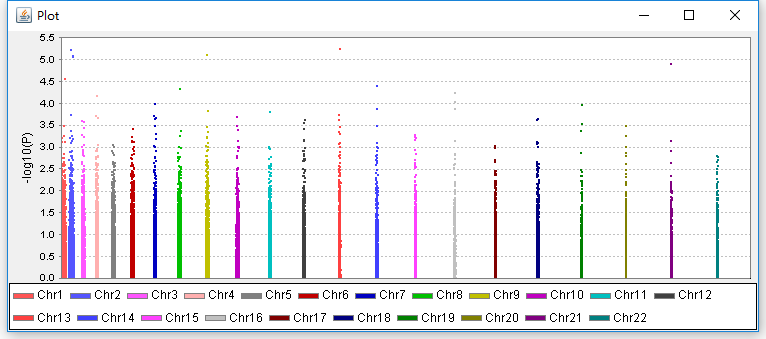
由于测试数据的SNP位点比较少,最后的效果图并不是特别美观。
扫描关注微信号,更多精彩内容等着你!











