欢迎关注”生信修炼手册”!
在所有的基因型填充软件中,都会区分常染色体和X染色体,分别进行填充,为何对于X染色体要单独处理呢?
众所周知,性染色体在男性中为XY, 女性为XX。对于X染色体而言,男性为单倍体,女性为二倍体。在进行分型时,是不是意味着男性X的分型结果都为纯合呢?
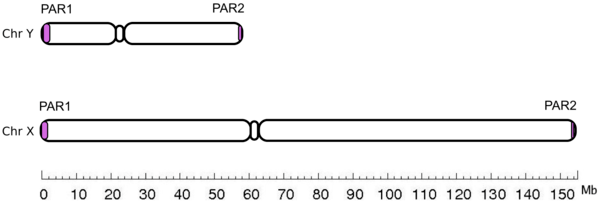
实际情况没有这么简单,在X染色体上存在了一些称之为PAR的特殊区域,图示如下
PAR全称如下
pseudoautosomal region
表示的是X和Y染色体间的同源区域,这些区域基因的遗传模式和常染色体类似,可以看作是二倍体。在hg38版本中,PAR区域对应的染色体位置如下
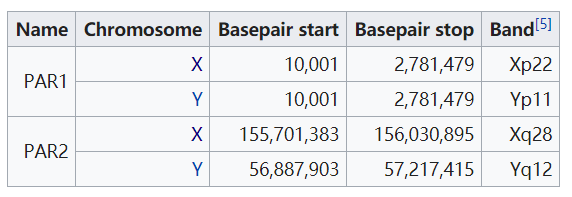
hg19版本中PAR区域对应的染色体位置如下
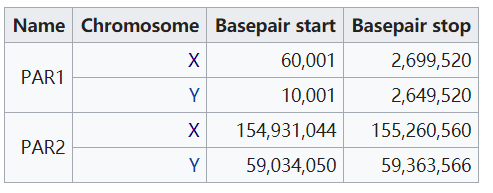
X染色体上有两段PAR区域,第一段位于头部,长度为2.6M左右, 第二段位于尾部,长度为320kp左右。
对于X染色体的基因型填充而言,PAR区域和非PAR区域需要单独处理。PAR区域在所有样本中都可以看作二倍体来处理,而非PAR区域在男性中为单倍体,女性中为二倍体,针对不同的性别需要分开处理。
在minimac中对于X染色体的基因型填充最能体现X染色体填充的复杂性,过程如下
1. Split the data into PAR and non-PAR
将PAR区域和非PAR区域分开,代码如下
# 根据非PAR区域的染色体位置进行提取
vcftools \
--gzvcf gwas.data.vcf.gz \
--chr X \
--from-bp 2699520 \
--to-bp 154931043 \
--recode \
--out Non.PAR.gwas.data
# 剔除非PAR区域,保留PAR区域的位点信息
vcftools \
--gzvcf gwas.data.vcf.gz \
--exclude-positions Non.PAR.gwas.data.recode.vcf \
--recode \
--out PAR.gwas.data2. Split the non-PAR data by Sex
对于非PAR区域, 将男性样本和女性样本分开,代码如下
# 根据样本ID进行提取
vcftools \
--vcf Non.PAR.gwas.data.recode.vcf \
--keep male.sample.list \
--recode \
--out Male.Non.PAR.gwas.data
vcftools \
--vcf Non.PAR.gwas.data.recode.vcf \
--keep female.sample.list \
--out Female.Non.PAR.gwas.data3. pre-phasing and convert vcf
只有男性的非PAR区域的分型结果直接就是单倍型了,所以除了这个数据外,PAR区域和女性的非PAR区域的分型结果都需要进行单倍型分析,代码如下
# 对PAR区域进行pre-phasing
mach1 \
-d PAR.gwas.data.dat \
-p PAR.gwas.data.ped \
--rounds 20 \
--states 200 \
--phase \
--interim 5 \
--prefix PAR.gwas.data.Phased.Output
# 格式转换
mach2VCF \
--haps PAR.gwas.data.Phased.Output.hap \
--snps PAR.gwas.data.Phased.Output.snps \
--prefix Phased.PAR.gwas.data
# 对女性的nonPAR区域进行pre-phasing
mach1 \
-d Female.Non.PAR.gwas.data.dat \
-p Female.Non.PAR.gwas.data.ped \
--rounds 20 \
--states 200 \
--phase \
--interim 5 \
--prefix Female.Non.PAR.gwas.data.Phased.Output
# 格式转换
mach2VCF \
--haps Female.Non.PAR.gwas.data.Phased.Output.hap \
--snps Female.Non.PAR.gwas.data.Phased.Output.snps \
--prefix Female.Non.PAR.gwas.data4. imputation
针对3种结果分别进行填充,代码如下
# Phased All Samples (PAR)
Minimac3 \
--refHaps refPanelChrX.Auto.vcf \
--haps Phased.PAR.gwas.data.vcf \
--prefix testRun.All.PAR
# Phased Female Samples (Non-PAR)
Minimac3 \
--refHaps refPanelChrX.Non.Auto.vcf \
--haps Phased.Female.Non.PAR.gwas.data.vcf \
--prefix testRun.females.Non.PAR
# Haploid Male Samples (Non-PAR)
Minimac3 \
--refHaps refPanelChrX.Non.Auto.vcf \
--haps Male.Non.PAR.gwas.data.recode.vcf \
--prefix testRun.males.Non.PAR详细步骤参考以下链接
https://genome.sph.umich.edu/wiki/Minimac3Cookbook:_Chromosome_X_Imputation
在impute2和Beagle中,则对上述步骤进行了封装,通过特定的参数来进行X染色体的填充,细节请参阅官方的说明文档。
由于X染色体在不同性别中的分布以及PAR区域的存在,针对X染色体的基因型填充需要单独处理。
·end·
—如果喜欢,快分享给你的朋友们吧—
扫描关注微信号,更多精彩内容等着你!











