欢迎关注"生信修炼手册"!
分析体细胞突变时,通常采用tumor_vs_nomal 的实验设计。在检测时,由于同时会检测出生殖细胞突变和体细胞突变,需要做的就是去除生殖细胞突变位点,那么剩下的就是体细胞突变位点了,GATK4 采用Mutect2 检测体细胞突变,分析流程如下: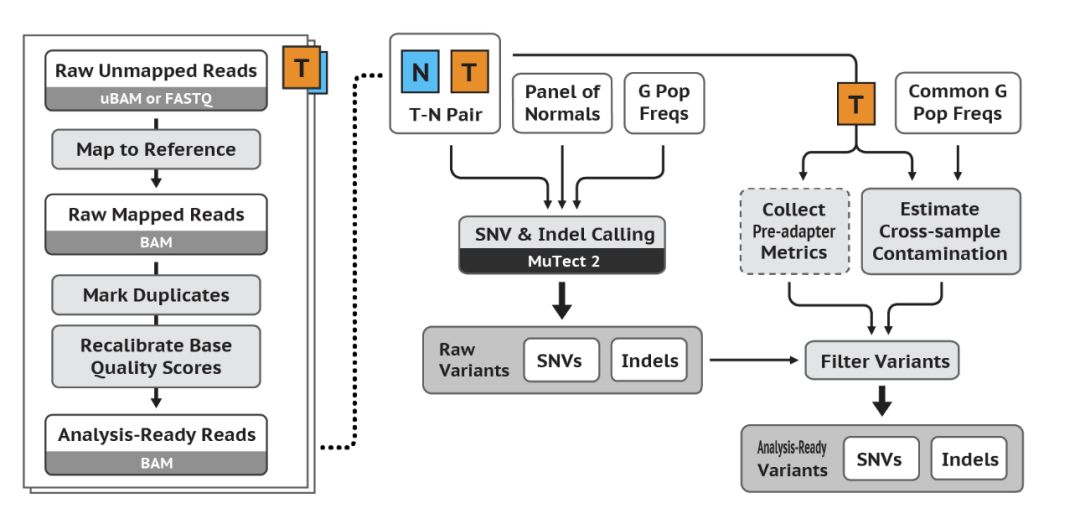
1. 根据normal 样本得到 panel of normal
首先对每个normal 样本,运行Mutect2
gatk Mutect2 \
-R reference.fa \
-I normal1.bam \
-tumor normal1_sample_name \
--germline-resource af-only-gnomad.vcf.gz \
-O normal1_for_pon.vcf.gz然后使用CreateSomaticPanelOfNormals命令创建panel of normal
gatk CreateSomaticPanelOfNormals \
-vcfs normal1_for_pon_vcf.gz \
-vcfs normal2_for_pon_vcf.gz \
-vcfs normal3_for_pon_vcf.gz \
-O pon.vcf.gz2. normal_vs_turmor 得到体细胞突变
命令如下:
gatk Mutect2 \
-R reference.fa \
-I tumor.bam \
-tumor tumor_sample_name \
-I normal.bam \
-normal normal_sample_name \
--germline-resource af-only-gnomad.vcf.gz \
--af-of-alleles-not-in-resource 0.00003125 \
--panel-of-normals pon.vcf.gz \
-O somatic.vcf.gzmutect2检测时,是成对检测的,需要一个normal bam 和 turmor bam, germline-resource指定一个生殖细胞突变的vcf文件,这里选择的是gnomAD数据库 ,链接如下
http://gnomad.broadinstitute.org
这个数据库收集了大量外显子和全基因组测序的SNP calling结果。af-of-alleles-not-in-resource指定germline-resource 变异位点的频率,低于该频率的位点认为是一个不可靠的生殖细胞突变位点。panel-of-normals指定第一步生成的pon.vcf.gz文件。
3. 过滤VCF文件
第一步,运行GetPileupSummaries
gatk-launch GetPileupSummaries \
-I tumor.bam \
-V small_exac_common_3.vcf \
-O pileups.table第二步,运行CalculateContamination
gatk-launch CalculateContamination \
-I pileups.table \
-O contamination.table第三步,运行FilterMutectCalls
gatk FilterMutectCalls \
-V somatic.vcf.gz \
-contamination-table contamination.table \
-O filtere扫描关注微信号,更多精彩内容等着你!











