前言
生物信息学研究中生物信息扮演着重要的角色。其中基因表达矩阵数据使用的频率非常的高,本文介绍如何在GEO数据库中获取相关的基因表达数据并做相关处理,这里以肺癌为例。
一、数据获取
1.通过NCBI官网进入GEO(https://www.ncbi.nlm.nih.gov/);
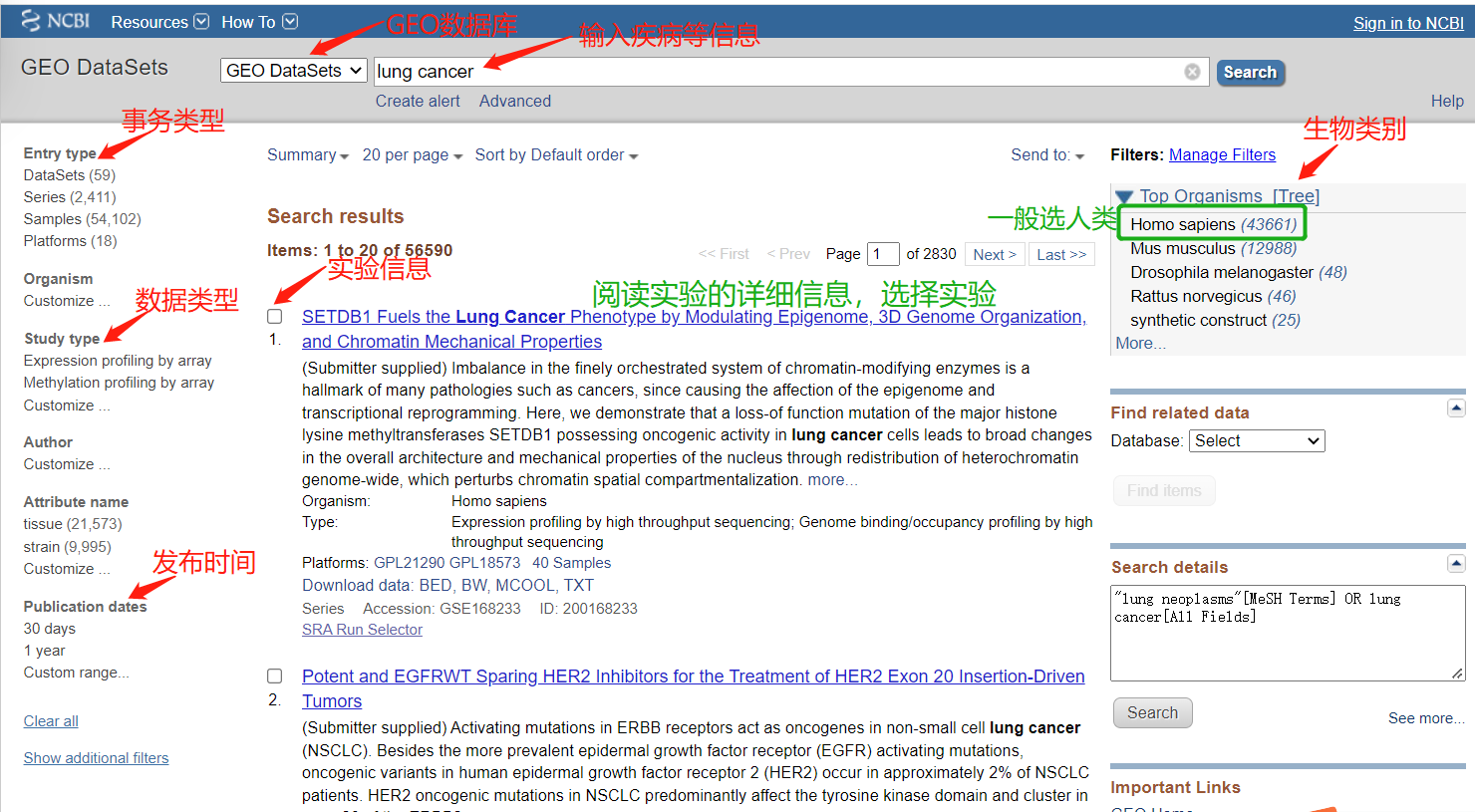
2.实验的详细信息:这部分主要关注平台信息,样本信息和表达数据
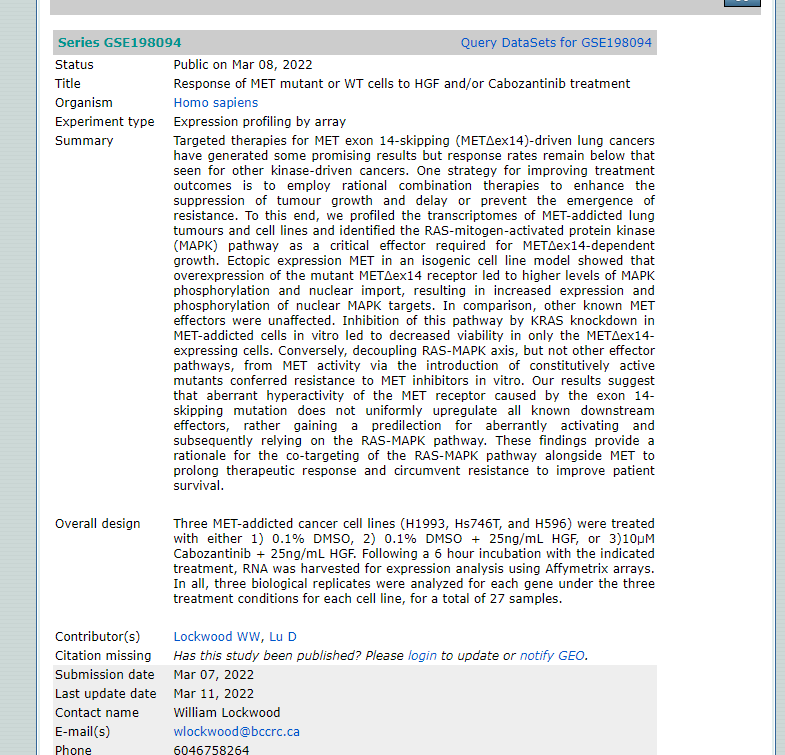
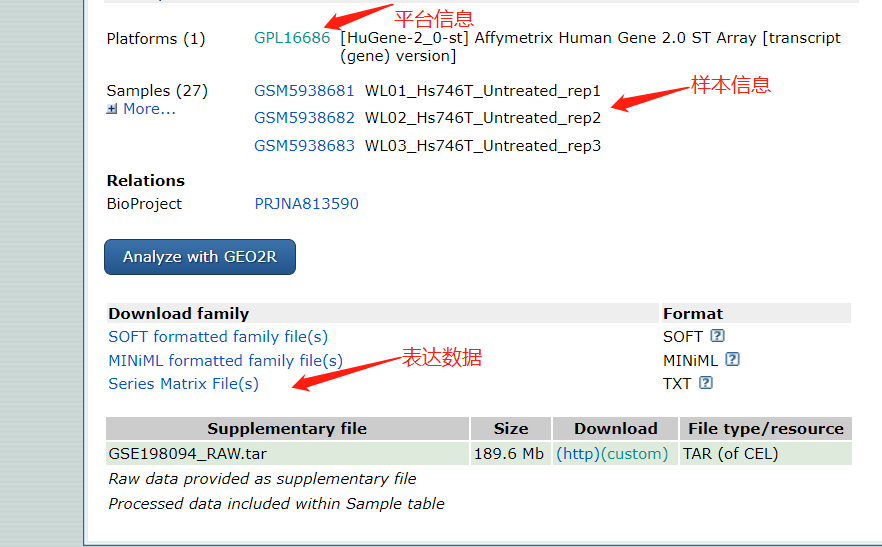
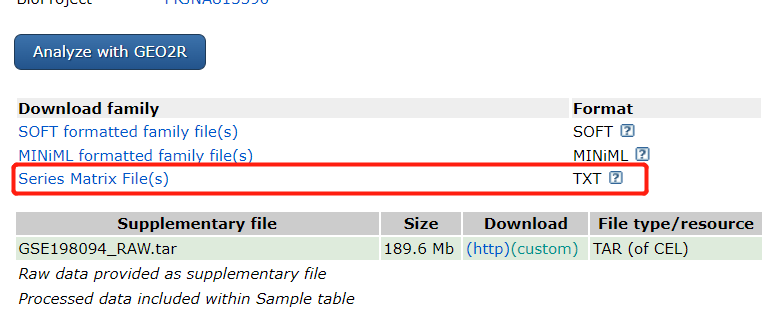
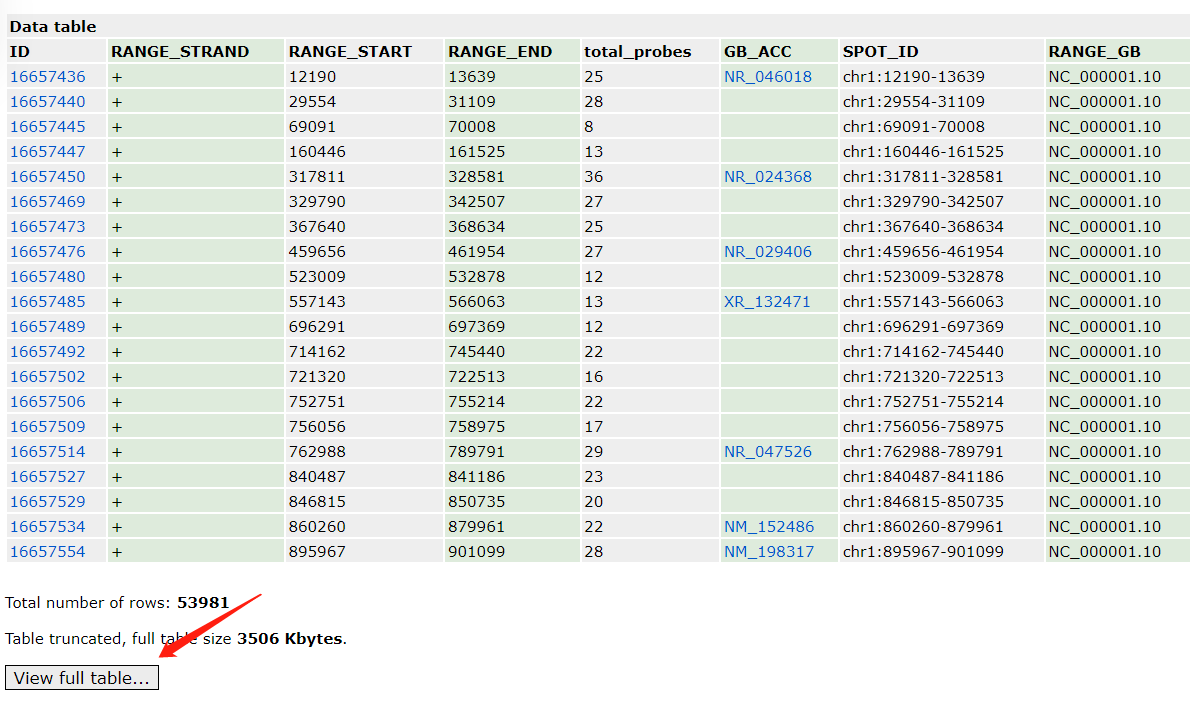
3.下载相关数据时,可以直接在这里下载,分别为样本表达数据和平台信息数据,在网站中分别为Series Matrix File和平台信息里的table
二、数据处理
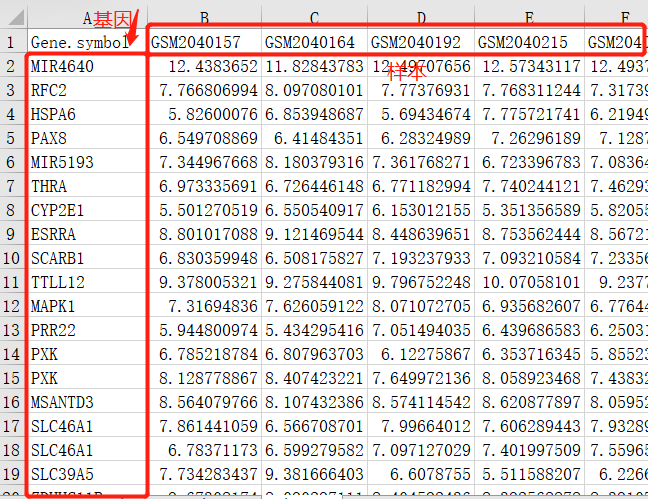
1.下载样本的探针的表达数据和平台中探针和基因的对应信息后,需要通过脚本或者EXCEL处理得到基因的表达数据,后期还要处理数据中的冗余数据等十分的麻烦,这里不详细介绍,还是推荐利用R语言直接获取,具体方法见R语言获取GEO表达数据。

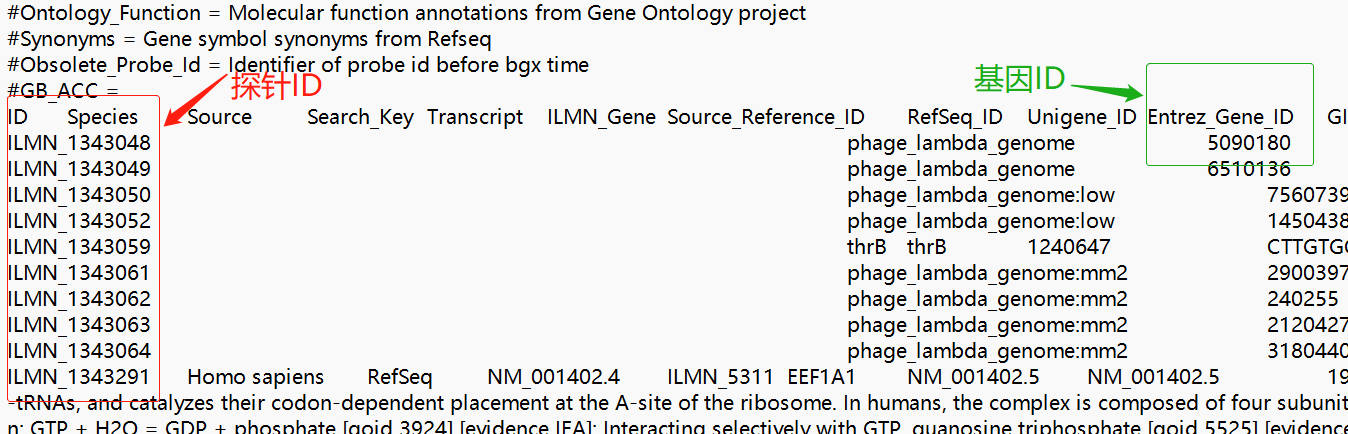
2.数据中存在多个探针对应一个基因的情况,故需要一个基因的所有表达数据统计出来作为该基因的表达水平,一般取均值,当然也可以根据需要取最大(最小)值。这一功能利用简单的python代码实现。代码和数据如下。
import pandas as pd
df = pd.read_excel('expree_data.xlsx', sheet_name='Sheet1')
df1 = df.groupby('Gene.symbol').mean() #
df1.to_excel('expree_data_x.xlsx')